scikit-learnのcross_val_scoreを使って交差検証(Cross Validation)をする方法
機械学習モデルを作る時、与えられたデータを全て用いてモデルの学習・精度向上を行うと、そのデータに対してのみ精度の良いモデル(理想のモデルに近づけていない。)が出来上がってしまい、未知のデータに対して適用できなくなってしまいます。
そのため通常、データをあらかじめ学習用と検証用に分けておき、学習用データでモデル作成→検証用データでモデルの精度を確かめるという手順でモデリングを進めていきます。

さて、上記のように学習データ内で精度の良いモデルを作るのですが、こちらも学習データに特化したモデルを作ってしまうと、いつまでたっても精度の良いモデルができません。(特に学習データが少ない場合。)
この問題を解決する手法が交差検証(Cross Validation)です。今回は交差検証の中でも、K-分割交差検証(k-Fold cross validation)について説明します。
K-分割交差検証では学習データをさらにk個に分割して学習用と検証用に分けて学習→検証を行うのですが、この時に検証データを全ての分割したデータに割り当てます。言葉で説明しにくいので下図参照。
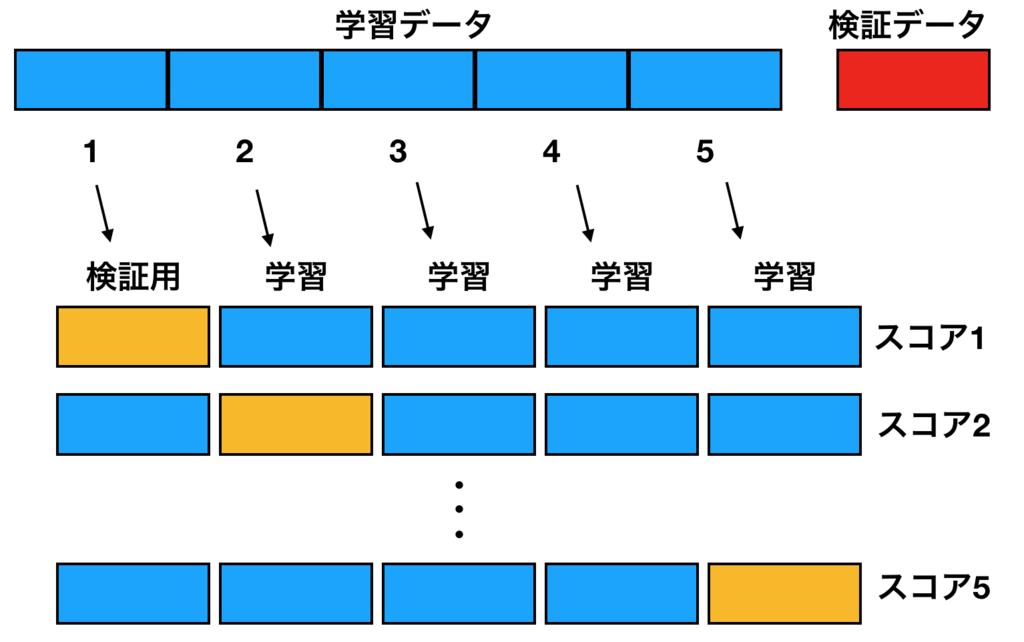
図の中では学習データを5分割しているのでk=5となります。検証データセットを図のように順にずらしていくことで各パターンのモデル精度をスコアにしてスコアの平均がもっとも良いモデルを採用→最初に分けた検証データで評価という流れです。
実際にやってみましょう。scikit-learnのcross_val_scoreという最強のツールを使えば一発でできてしまいます。
まずは、以下をあらかじめインポート。
サンプルデータとして乳がんのデータを使います。
# scikit-learnの入っている乳がんのデータ from sklearn.datasets import load_breast_cancer # 交差検証 from sklearn.model_selection import cross_val_score # ロジスティック回帰 from sklearn.linear_model import LogisticRegression # SVM from sklearn.svm import LinearSVC # 決定木 from sklearn.tree import DecisionTreeClassifier # k-NN from sklearn.neighbors import KNeighborsClassifier # ランダムフォレスト from sklearn.ensemble import RandomForestClassifier
データの中身を見ておきましょう。cancer.dataが説明変数、cancer.targetが目的変数となります。
説明変数の数がとても多いのですね。下の図では見えないですが、目的変数は乳がんかどうかといういみで0,1のデータになっています。
cancer = load_breast_cancer()
df = pd.DataFrame(cancer.data, columns=cancer.feature_names)
df['target'] = cancer.target
df.describe()
output>

次にモデリングと交差検証によるスコアリングを一気に行います。
こんなに少ないコードでできてしまうからすごい。。
ロジスティック回帰、SVM、決定木、K近傍法、ランダムフォレストでモデリングを行い比較します。
交差検証は→cross_val_score(モデルのクラス、説明変数、目的変数、分割数(今回は5分割))
これで記述できます。dfには交差検証によるスコアが各モデル5個ずつできます。
models = [LogisticRegression(),LinearSVC(),DecisionTreeClassifier(),KNeighborsClassifier(n_neighbors = 6),RandomForestClassifier()] scores = {} for model in models: scores[str(model).split('(')[0]] = cross_val_score(model, cancer.data, cancer.target, cv=5) df = pd.DataFrame(scores)
最後にモデルごとにスコアの平均を取って、精度の良いモデルを採用します。
df.mean()
output>

とりあえず、ランダムフォレストが一番良さそうです。